Motivation
- Eine grundlegende
Aufgabe in der Molekularbiologie
ist die Analyse von menschlichen Chromosomen. Ein Chromosom besteht aus
einer Aneinander-reihung von etwa 100 Millionen Basenpaaren. Eine
Rekonstruktion dieser Reihenfolge direkt aus einem kompletten Chromosom
ist zur Zeit nicht möglich.
- Daher
ist es notwendig, den vollständigen DNA-Strang in kleinere Teile zu
zerlegen und diese Fragmente einzeln zu untersuchen.
- Das
Problem dabei ist aber, dass bei der Zerlegung Informationen über die
An-ordnung der DNA-Fragmente (Clones)
verloren gehen. Das Problem der Wieder-herstellung dieser Ordnung nennt
man Physical Mapping.
Vorgehensweise
- Eine
mögliche Vorgehensweise beim Physical Mapping arbeitet mit sogenannten STSs (Sequence Tagged Sites),
auch Probes genannt. Das sind
DNA-Sequencen, die nur an einer einzigen Stelle des Chromosoms
auftreten. Diese Probes können z.B. durch Extrahieren von DNA-Sequenzen
aus den Enden der Clones gewonnen werden, in diesem Fall spricht man vom
Physical Mapping Problem mit
Endprobes.
- Mit
Hilfe von Hybridisierungsexperimenten
kann man zu jedem Probe-Clone-Paar
feststellen, ob das Probe innerhalb der Clone-Sequenz liegt, oder nicht.
Normalerweise hybridisiert das Probe mit dem Clone, falls es im Innern
liegt, ansonsten nicht.
- Die
Ergebnisse dieser Experimente lassen sich also als eine Matrix mit
#Clones Zeilen und #Probes Spalten schreiben (#Clones bzw. #Probes steht
dabei für Anzahl der Clones bzw. Probes). Die Einträge dieser Hybridisierungsmatrix sind 1 (in
Abb. 1 und 2 ausgefüllt dargestellt), falls das entsprechende
Testergebnis positiv ist (Probe und Clone hybridisieren) und 0 (in Abb.
1 und 2 nicht ausgefüllt dargestellt) bei einem negativen Ergebnis.
- Aus
dieser Matrix soll nun die Anordnung der Clones im Chromosom
rekonstruiert werden.
Fehlerbehandlung
- Das
wesentliche Problem bei dieser Rekonstruktion ist, dass die
Hybridisierungsexperimente fehlerbehaftet sind. Zwei der Fehler, die
typischerweise auftreten, sind zum einen die false-positive Fehler (eine Hybridi-sierung wird angezeigt,
obwohl das Clone das Probe nicht enthält und zum anderen die false-negative Fehler (keine
Hybridisierung wird angezeigt obwohl das Probe in Wirklichkeit innerhalb
des Clone liegt).
- Ohne
diese Fehler könnte das Physical Mapping Problem einfach auf das Lösen
eines sogenannten Consecutive
Ones Problem zurückgeführt werden.
- Will man
aber die tatsächliche Anordnung der Probes trotz Fehler möglichst exakt
bestimmen, so greift man auf die Idee der Maximum-Likelihood-Schätzung zurück. D.h. man versucht aus
einer beobachteten Hybridisierungsmatrix die eigentliche Matrix (die im
fehlerfreien Fall entstanden wäre) zu schätzen. Kennt man nämlich die
Wahrscheinlichkeiten für das Auftreten von false-positive- und
false-negative-Fehlern, so kann man zu jeder zulässigen Matrix (zulässig
bedeutet, dass diese Matrix im fehlerfreien Fall hätte erzeugt werden
können) die bedingte Wahrscheinlichkeit dafür berechnen, dass diese
Matrix die eigentliche Matrix ist, unter der Bedingung der tatsächlich
beobachteten Hybridisierungsmatrix.
Optimierungsproblem
- Man
möchte diejenige Matrix finden, für die diese Wahrscheinlichkeit maximal
ist. Dazu muss man ein sogenanntes kombinatorisches
Optimierungsproblem lösen. In diesem Fall ein gewichtetes Consecutive Ones Problem.
Branch-and-Cut
- Dieses
Optimierungsproblem lösen wir mit dem sogenannten Branch-and-Cut Ansatz. Dabei wird ein Polytop konstruiert,
dessen Ecken zu den zulässigen Lösungen des Problems gehören. Gesucht
wird dann die optimale Ecke im Polytop.
- Der
Branch-and-Cut Ansatz bietet den großen Vorteil, dass man damit nicht
nur sehr gute Lösungen finden kann, sondern auch zu jeder Zeit in der
Optimierungsphase eine Abschätzung erhält, wie weit die bis dahin beste
Lösung höchstens vom Optimum entfernt ist.
|
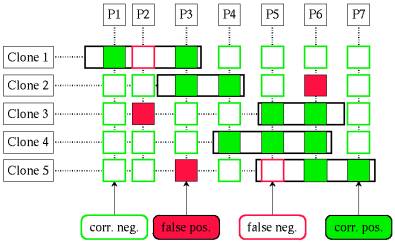
Abb. 1: Dieses Bild zeigt eine Hybridisierungsmatrix für das
Physical Mapping Problem ohne End-Probes. Ausgefüllte Quadrate zeigen eine
Hybridisierung an, entsprechend zeigen nicht ausgefüllte Quadrate an, dass
das Probe nicht mit dem entsprechenden Clone hybridisiert hat. Weiter sind
fehlerhafte Einträge (false-positives und false-negatives) rot und korrekte
Einträge grün dargestellt. In diesem Fall kann man der Zeichnung leicht die korrekte
Anordnung der Probes und damit auch der Clones entnehmen, man beachte aber,
dass die Information, welche Einträge fehlerbehaftet sind und welche nicht,
in der Praxis nicht vorliegt.
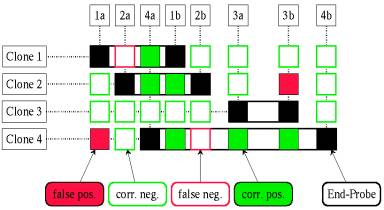
Abb. 2: Hier nun
eine Hybridisierungsmatrix für das Physical Mapping Problem mit End-Probes.
Der Unterschied besteht darin, dass hier die Probes von DNA-Sequenzen aus den
Enden der Clones gewonnen werden. Ein Hybridisierungstest zwischen Clone und
den beiden zugehörigen End-Probes ist daher nicht notwendig (entsprechenden
Quadrate sind schwarz dargestellt), da die relative Lage in beiden Fällen
bereits bekannt ist.
Ansprechpartner
Marcus Oswald
Im Neuenheimer Feld 368
69120 Heidelberg
Telefon: 06221/54-5746
Email: Marcus.Oswald@informatik.uni-heidelberg.de
|


![]()

